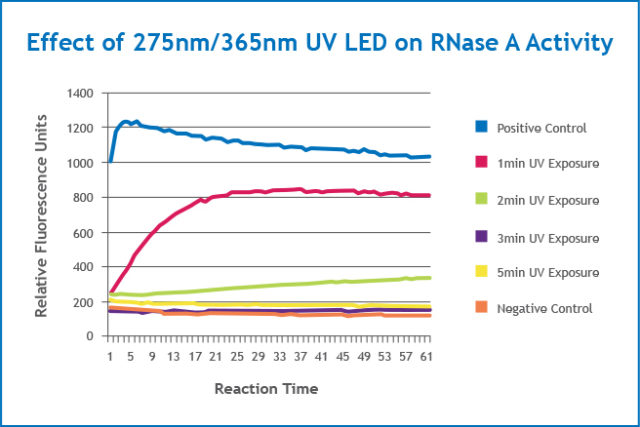
Die UV-LED-Technologie bietet eine neue Methode zur Dekontamination von Ribonuklease (RNase) für Sequenzierlabore der nächsten Generation. Die UV-Dekontamination von Ribonuklease mit hoher Intensität ermöglicht es Forschern, viel Zeit und Geld zu sparen und gleichzeitig konsistente, genaue Ergebnisse zu gewährleisten.
Wie Wissenschaftler bereits wissen, Laborkontamination ist ein andauerndes Problem für Experimente, die im Next-Generation Sequencing (NGS) durchgeführt werden. Signifikante Mengen an RNase-Kontamination in der Umgebung können innerhalb von Minuten auftreten. Verunreinigungen wie RNase A sind ohne scharfe Chemikalien nur schwer irreversibel zu inaktivieren.
Phoseon's KeyPro™ Dekontaminations-System eignet sich besonders für Abs, die eine hochreine RNA-Bibliotheksvorbereitung, Sequenzierung und PCR benötigen. Unter Verwendung von Phoseons patentierter Halbleiter-Lichtmatrix (SLM)™ -Technologie ermöglicht es dem KeyPro-System, die Produktivität im Labor zu erhöhen. Die vollständige Inaktivierung von Laborkontaminationen, einschließlich der schwer abzutötenden RNase A, kann mit dem KeyPro-System in weniger als fünf Minuten und zu einem Bruchteil der Kosten herkömmlicher Methoden durchgeführt werden. Laden Sie Reagenzien und Lösungsmittel und führen Sie Ihren Dekontaminationszyklus kurz vor der Probenzugabe durch.
Warum Ribonuklease-Kontamination ein Problem für RNA-Sequenzierlabore ist
In einer Laborumgebung ist der wichtigste Aspekt von RNA-Protokollen die Isolierung und Erhaltung von RNA in voller Länge, die nicht abgebaut wurde, für die Analyse oder die Verwendung als Reaktionssubstrat. Dieser Prozess wird durch das Vorhandensein von RNase behindert. Ob bei der Vorbereitung von Gesamt-RNA-Bibliotheken für Next Generation Sequencing (NGS) oder bei der Untersuchung einzelner RNAs (iCLIP), der Abbau durch RNasen ist ein wiederkehrendes Problem im Labor, das verschiedene Reinigungsmethoden erfordert.
Sobald eine Packung mit Einwegpipetten geöffnet wird, kann der Inhalt kontaminiert werden und ist nicht mehr für die RNA-Arbeit geeignet. Pipetten, die auf dem Arbeitstisch liegen, können Staub und mikrobielle Verunreinigungen aus der Raumluft ansammeln und müssen häufig nachgereinigt werden. Die Reinigung von Oberflächen und Geräten mit Sprays und Spülungen kann chemische Rückstände hinterlassen, eine weitere Art der Kontamination, die nachgeschaltete biochemische Reaktionen stören kann.
Außerdem kann wiederholter Kontakt mit Reinigungslösungen oder Einweichen Metall korrodieren oder Kunststoffoberflächen zersetzen. Wie sauber ist sauber genug? Sauber genug ist, wenn Sie keine langwierigen Protokolle aufgrund von degradierter RNA wiederholen müssen. Selbst Spuren von RNase haben aufgrund ihrer katalytischen Wirkung einen großen Einfluss auf die RNA-Sequenzierung. Nicht sauber führt zu Zeit- und Geldverlusten.

Erfahren Sie mehr über das KeyPro Dekontaminationssystem für Next Generation Sequencing (NGS)